沖縄科学技術大学院大学と日本医療研究開発機構,AIが脳のマッピング研究を加速
—日本の脳科学プロジェクトが AIにより脳回路マッピング技術の精度と信頼性を向上—
2020-12-18
沖縄科学技術大学院大学(OIST)が,理化学研究所脳科学センターおよび京都大学と共同で, MRIによる脳の神経経路の解析の精度を向上させる新たな手法を開発した。この手法はパーキンソン病やアルツハイマー病などの精神神経疾患に伴う神経経路の変化の解明に貢献する。本研究成果は, 12月18日日本時間 19時(英国時間 10時)に科学誌 Scientific Reportsに掲載される。
1.背景
OISTの神経計算ユニットを率いる銅谷賢治教授は,研究の背景について,「脳のさまざまな領域の間の接続の全容を明らかにすることを “コネクトーム ”と呼ぶが,これは脳が実現する複雑な機能を完全に理解するためには不可欠です」とコネクトームの重要性を説明。
コネクトームを明らかにするためには,脳全体に広がる神経線維を追跡する必要がある。動物では,脳内の複数のポイントに蛍光トレーサーを注入し,これらのポイントから伸びる神経線維の経路を画像化することができる。しかしこのやり方では,一つの注入点につき何百もの脳切片を分析し,さらにこれを,注入点を変えた多数の脳に関して行う必要がある。このやり方を人間に応用することは不可能である。
しかしながら,磁気共鳴画像(MRI)の技術が進歩したことで,脳を解剖することなくコネクトームを推定することが可能になった。拡散MRI神経線維追跡と呼ばれる手法は,強力な磁場の中でコンピューターアルゴリズムで脳の各点での線維の向きを接続することで,脳全体の神経線維の経路を推定します。(画像1)
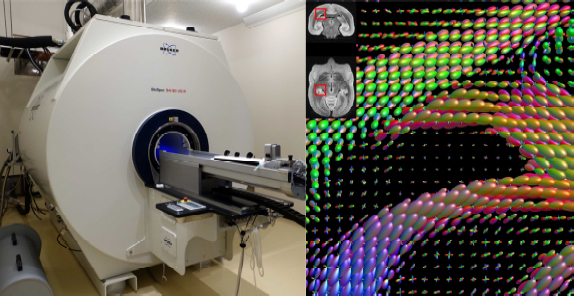
画像1:理化学研所のMRIスキャナーと拡散MRIデータ
(左)研究に使用した理化学研究所脳科学センターのMRIスキャナー。水分子の拡散する方向を解析することで,脳を解剖することなく神経結合を推定することができる。(右)拡散MRIは脳の各部での水の拡散方向を計測する。この図は脳の一部を拡大し,そこでの水の拡散の方向を楕円体で示している。この情報をもとに神経線維追跡アルゴリズムにより神経線維の経路を推定する。
提供: (左)理化学研究所脳科学センター畑純一,岡野栄之博士。(右)MRtrix viewer 3.0.1 により作成。
ただ,現在の推定アルゴリズムで得られる結果は確信を持てるものではない。例えば,カメラの設定の仕方によって写真が異なって見えるのと同様に,アルゴリズムに研究者が選択した設定(パラメータ)次第で,非常に異なるコネクトームが生成される。
「この方法の信頼性には大きな懸念があります。不正確な線維追跡によって,実際には存在しない神経接続を示してしまう可能性があるのです」と,本研究の筆頭著者で神経計算ユニット所属のポストドクトラルスカラー,カルロス・グティエレツ博士は述べている。
さらに現在のアルゴリズムでは,脳の離れた領域の間をつなぐ神経線維を検出するのは困難です。しかし,これらの長距離接続は,脳がどのように機能するかを理解するために最も重要なものでもあります」と,グティエレツ博士は説明する。
2013年に日本の「革新的技術による脳機能ネットワークの全容解明プロジェクト Brain / MINDS」が立ち上がり,人間に近い脳構造を持つ小型霊長類であるマーモセットの脳をターゲットとした系統的なデータ取得が始まった。
同プロジェクトは,マーモセット脳のコネクトームを作成するために,脳を解剖する侵襲的手法と MRIを利用した非侵襲的手法の両方を用いている。マーモセットの脳を解剖して蛍光トレーサーの撮像をする前にその脳のMRI画像を取得する。
「このプロジェクトで得られるデータは,同じ脳から2つの手法により得られる結果を比較し,拡散MRI神経線維追跡アルゴリズムがより正確な結果を与えるようにパラメータを決定するという,他に例のない研究を可能にしたのです」とグティエレツ博士は述べている。
本研究では,広く使用されている 2つの異なる神経線維追跡アルゴリズムのパラメータの最適化を行った。長距離の結合をなるべく多く検出しつつ,実際には存在しない神経線維の検出を最小限に抑えることを目標にした。
2.研究手法・成果
研究チームは,さまざまなパラメータの組み合わせを手作業で試す代わりに, AIに目を向け「進化的アルゴリズム」と呼ばれる手法を使用した。
今回用いたアルゴリズムは生物の進化のしくみにならった最適化手法で,ランダムな変化を加えた複数のパラメータを用意し,それらを使って神経線維追跡アルゴリズムにより拡散MRIデータからコネクトームを推定する。その結果を蛍光トレーサーによって検出された結合と比較することで評価し,より評価の高いパラメータを選択するという手順を繰り返す。
検証実験では 10頭のマーモセットの脳からの蛍光トレーサーと MRIのデータを最適化に使用した。
しかしAIを用いても,最適なパラメータを選択することは容易ではなかった。「一部のパラメータは誤検出率を低くすることができるかもしれませんが,長距離接続の検出が難しくなります。したがって,どのパラメータを選択するかを決定するには,常にトレードオフが必要です」と,グティエレツ博士は説明する。
この進化的アルゴリズムを複数の脳に対して繰り返しながら,個々の脳で高い評価を得たパラメータを互いに交換し,異なる脳に対して共通に良く働くパラメータが選択されるようにした。最後に選択されたパラメータを平均して,異なる脳に共有して最適なパラメータを作成した。(画像2)
「パラメータの組み合わせは重要なステップでした。個々の脳は異なるため,特定の 1つの脳に最適なパラメータの独自の組み合わせが常に存在します。しかし私たちの目的は,すべてのマーモセットの脳にうまく機能する汎用性を持つ最良のパラメータのセットを見つけ出すことでした。」
最適化された共通パラメータを使用して,最適化には使わなかった別のマーモセットの脳でコネクトームの推定を行った場合に,標準パラメータを使うよりも正確なコネクトームが生成されることも確認しました。(画像2,3)
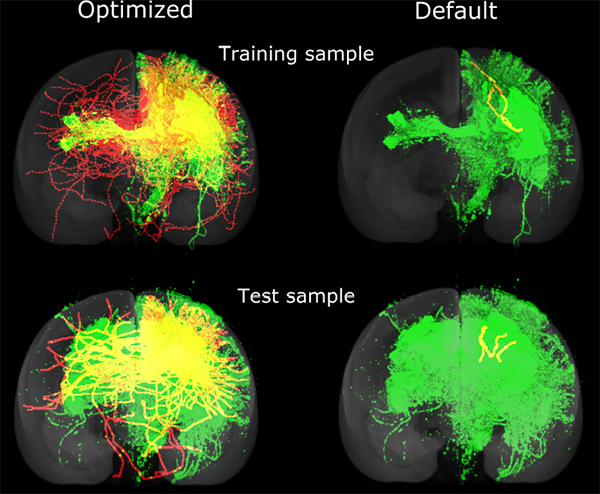
画像2:最適化されたパラメータと標準パラメータによる神経線維追跡結果の比較
緑色は脳の1点に蛍光トレーサーを注入して検出された神経線維を,赤色は拡散MRI神経線維追跡により推定された線維のうち,トレーサー注入部位につながるものを示す。両者が一致している部分は黄色で示される。最適化したパラメータによる神経線維追跡(左)では,標準的に使われるパラメータによる結果(右)より多くの,より遠くまでの線維が検出されている。上の例は最適化に使った脳であるが,下の例は最適化には使わなかった新たな脳サンプルに対しても追跡性能が向上(黄色の部分が増えている)ことを示す。
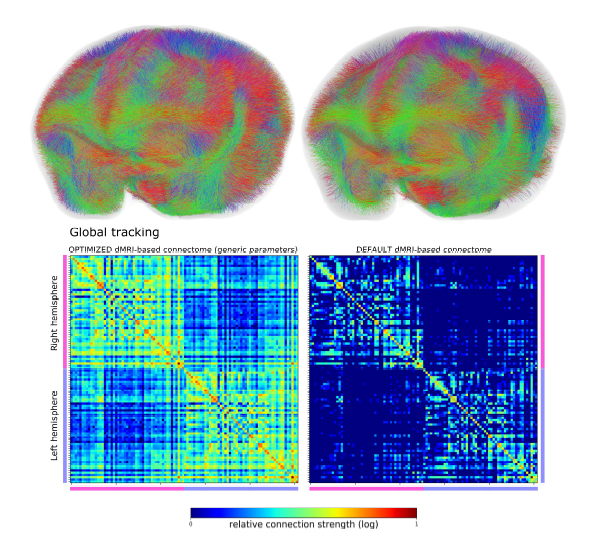
画像3:最適化したパラメータと標準パラメータでの全脳のコネクトームの違い。
(左上)最適化したパラメータにより拡散MRI神経線維追跡アルゴリズムにより推定された全脳のコネクトーム。(右上)同じアルゴリズムで標準のパラメータにより推定されたコネクトーム。より少ない線維しか見ることができない。(下)推定された脳の領野間の結合の強さを行列として表したもの。上と同様に左は最適化したパラメータ,右は標準のパラメータによるもの。
3.研究のインパクト,今後の予定
標準的に使われるパラメータと最適化されたパラメータによる神経線維追跡の結果に大きな違いが見られたことは,拡散 MRIによるコネクトーム研究に鋭い警鐘を発するものある。
「最適化または検証されていないアルゴリズムを使用した研究には疑問が投げかけられたことになります」とグティエレツ博士は警告する。
将来的にチームは,最適なパラメータを特定するプロセスを高速化するようにアルゴリズムを改善して,神経障害または精神障害に伴う脳のコネクトームの変化をより正確に検出できるようにしたいと考えている。
「最終的には,拡散 MRI神経線維追跡を使用して,人間の脳全体をマッピングし,健康な脳と病気の脳の違いを特定することができるはずです。これにより,これらの障害の治療方法の開発に一歩近づくことができます。」とグティエレツ博士は抱負を語っている。
4.研究プロジェクトについて
本研究は, AMEDの「革新的技術による脳機能ネットワークの全容解明プロジェクト」から資金提供を受けた。
<論文タイトルと著者>
タイトル: Optimization and Validation of Diffusion MRI-based Fiber Tracking with Neural Tracer Data as a Reference
著者: Carlos Enrique Gutierrez1*, Henrik Skibbe2, Ken Nakae3, Hiromichi Tsukada1, JeanLienard1, Akiya Watakabe4, Junichi Hata5,9,10, Marco Reisert6, Alexander Woodward7,Yoko Yamaguchi8,11,12, Tetsuo Yamamori4, Hideyuki Okano5,10, Shin Ishii3, and KenjiDoya1 *連絡先の著者
1Neural Computation Unit, Okinawa Institute of Science and Technology Graduate University, Okinawa, Japan 2Brain Image Analysis Unit, RIKEN Center for Brain Science, Wako, Japan 3Integrated Systems Biology Laboratory, Department of Systems Science, Graduate School of Informatics, KyotoUniversity, Kyoto, Japan 4Laboratory for Molecular Analysis of Higher Brain Function, RIKEN Center for Brain Science, Wako, Japan 5Laboratory for Marmoset Neural Architecture, RIKEN Center for Brain Science, Wako, Japan 6Department of Medical Physics, Medical Center, Freiburg University, Germany 7Connectome Analysis Unit, Riken Center for Brain Science, Wako, Japan 8Applied Electronics Laboratory, Kanazawa Institute of Technology, Japan 9Division of Regenerative Medicine, The Jikei University School of Medicine, Tokyo, Japan 10Department of Physiology, Keio University School of Medicine, Tokyo, Japan 11Graduate School of Information Science and Technology, The University of Tokyo, Tokyo, Japan 12Laboratory for Cognitive Brain Mapping, RIKEN Center for Brain Science, Wako, Japan
掲載誌: Scientific Reports DOI:10.1038/s41598-020-78284-4
●問い合わせ先
<研究に関する問い合わせ>
銅谷賢治(どうやけんじ)
沖縄科学技術大学院大学(OIST)神経計算ユニット教授
TEL 098-966-8628 E-mail: doya@oist.jp
<事業に関する問い合わせ先>
国立研究開発法人日本医療研究開発機構(AMED)疾患基礎研究部疾患基礎研究課
TEL 03-6870-2286 FAX 03-6870-2243 E-mail:brain-m@amed.go.jp
